Investigadores en Alemania han Desarrollado un Enfoque Prometedor Para Suprimir la Replicación Viral del Síndrome Respiratorio Agudo Severo Coronavirus 2 (SARS-CoV-2), el Agente Que Causa la Enfermedad Por Coronavirus 2019 (COVID-19 .
El equipo, de la Universidad Técnica de Berlín, la Universidad Libre de Berlín y el Centro Alemán de Investigación de Infecciones en Charitéplatz, Berlín, diseñó ocho pequeños ARN de interferencia (ARNip) que se dirigen a una región altamente conservada de SARS-CoV-2.
Como se informó en la revista Viruses , el más eficiente de estos ARNip, siCoV6, inhibió significativamente la replicación de los viriones del SARS-CoV-2 y fue activo contra la variante B.1.1.7 (alfa).
Curiosamente, siCoV6 Fue Incluso Muy Activo Contra un Miembro de la Familia del SARS-CoV-1, lo que Apunta a un ARNip Potente con Amplia Actividad Antiviral Contra los Virus del SARS-CoV.
Jens Kurreck y sus Colegas dicen Que esta Amplia Actividad Antiviral Convierte a siCoV6 en un Candidato Prometedor Para el Tratamiento de las Infecciones por Coronavirus del SARS.
.jpg)
Se necesita con urgencia el desarrollo de antivirales eficaces
Desde que comenzó el brote de COVID-19 a fines de diciembre de 2019, el agente causante del SARS-CoV-2 ha infectado a más de 238 millones de personas y se ha cobrado la vida de más de 4.86 millones.
A pesar del desarrollo y la aprobación exitosos de varias vacunas, las opciones de tratamiento para los pacientes con enfermedades graves son limitadas y se necesitan con urgencia medicamentos antivirales eficaces.
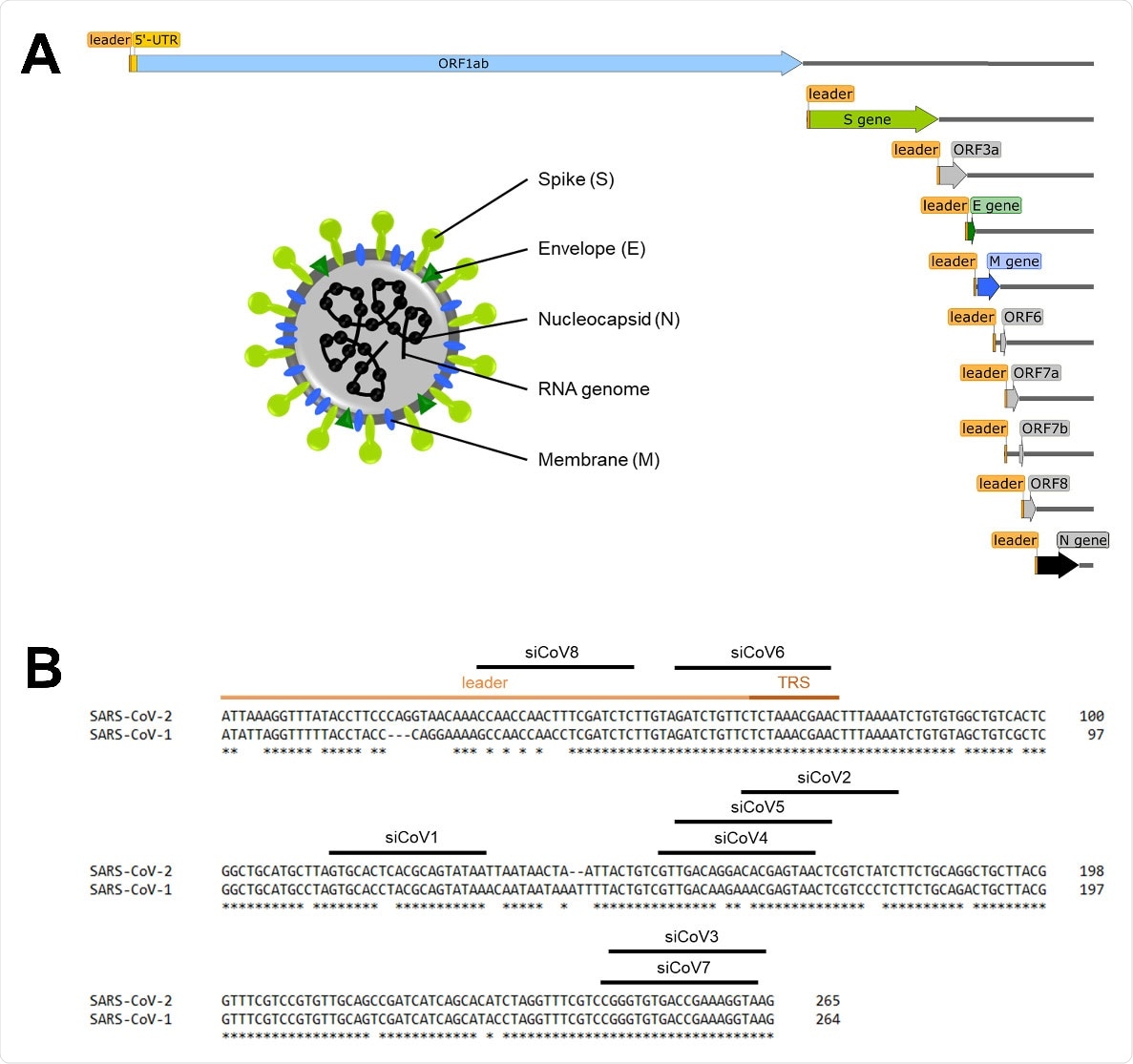
El genoma del ARN del SARS-CoV-2 tiene aproximadamente 30 kilobases de longitud y codifica 16 proteínas no estructurales (nsp1-16) que se requieren para la replicación del ARN.
Una vez que el SARS-CoV-2 ha entrado en una célula huésped, todas estas proteínas se traducen directamente e inducen la replicación del genoma del ARN.
Una característica única de los coronavirus es que no solo se produce el ARN genómico completo, sino también los ARN subgenómicos. Todos estos ARN subgenómicos comparten el mismo extremo 5 '(de alrededor de 75 nucleótidos), que se conoce como la "secuencia líder".
Un enfoque prometedor para inhibir la replicación viral
Un enfoque prometedor para inhibir la replicación viral es el uso de ARN de interferencia (ARNi), un proceso eficaz de silenciamiento génico postranscripcional que puede desencadenarse mediante ARNip. Este proceso conduce a la degradación del ARN mensajero (ARNm) de una manera específica de secuencia e inhibe la expresión génica.
El direccionamiento de la expresión génica a través de RNAi se está desarrollando como una nueva estrategia terapéutica, y previamente se han investigado numerosos agentes basados en RNAi para tratar infecciones virales. Los virus de ARN como el SARS ‐ CoV ‐ 2 son candidatos particularmente prometedores, ya que no solo sus ARNm, sino también su ARN genómico pueden ser dirigidos directamente.
En consonancia con esto, Kurreck y sus colegas y otros grupos de investigación han demostrado previamente que la replicación de virus de ARN como la influenza y la hepatitis C puede inhibirse eficazmente por ARNi en varios modelos de enfermedades celulares y animales.
¿Qué implicó el estudio actual?
El equipo diseñó y probó ocho ARNip dirigidos contra la región 5 'sin traducir (5'-UTR) del SARS-CoV-2.
La 5'‐ UTR es una región altamente conservada que es crucial para la replicación y transcripción del ARN viral, lo que la convierte en un objetivo prometedor para el diseño de tratamientos basados en ARNi.
Los ensayos informadores iniciales confirmaron que los ocho ARNip se dirigieron de manera eficiente al 5'‐ UTR del SARS ‐ CoV ‐ 2.
El candidato más prometedor del panel, siCoV6, apuntó a la secuencia líder que está presente tanto en el genoma viral como en todos los ARN subgenómicos que son esenciales para la replicación del virus .
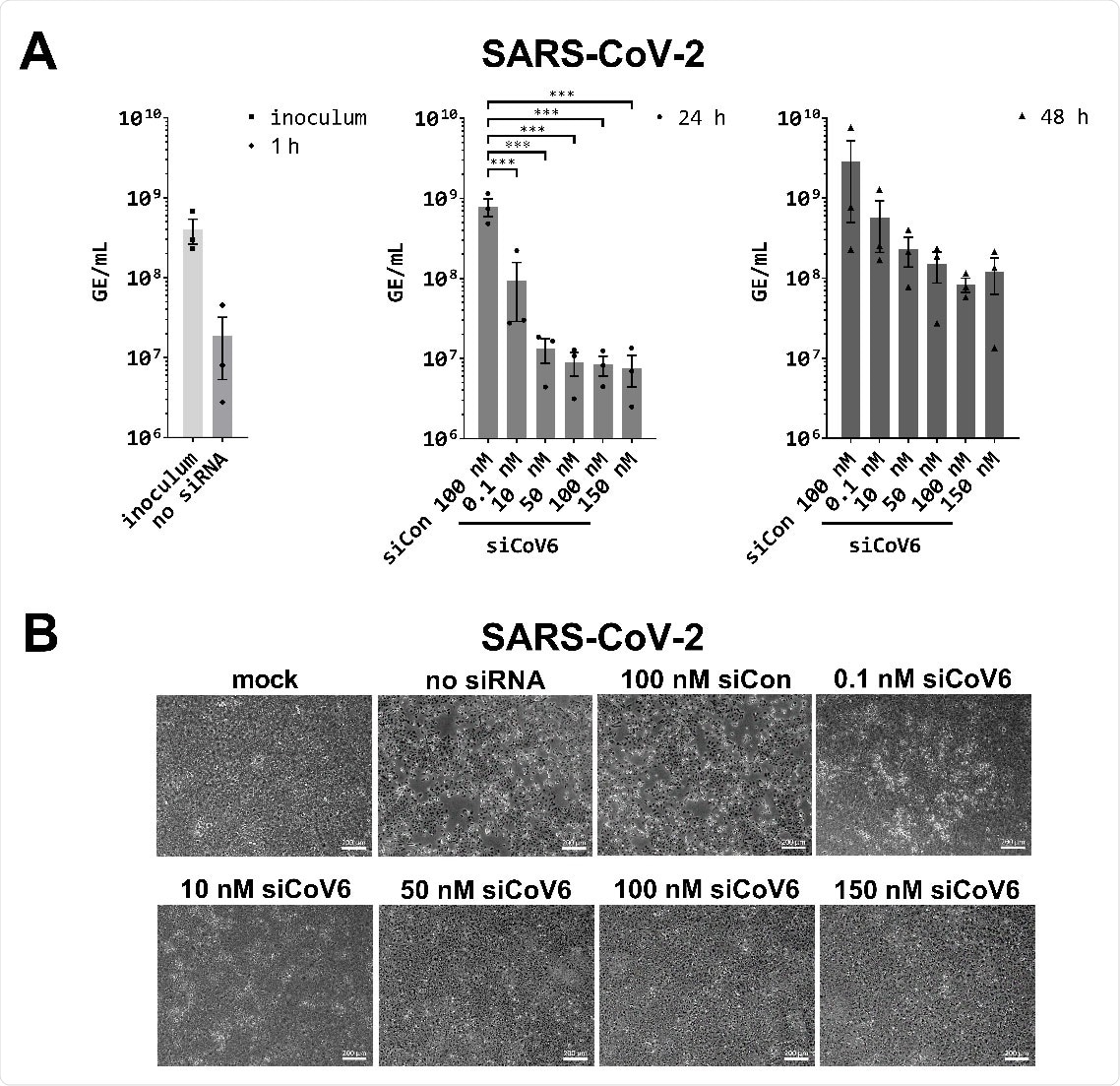
Cuando se probó con SARS-CoV-2 infeccioso, siCoV6 inhibió eficazmente la replicación del virus en las células Vero E6 en dos órdenes de magnitud (99%) a solo ARNip 10 nM y protegió a las células del efecto citopático.
Además, dado que su secuencia diana está muy conservada, siCoV6 también mostró una actividad inhibidora prometedora contra la variante alfa del SARS ‐ CoV ‐ 2 e incluso contra un miembro de la familia del SARS ‐ CoV ‐ 1.
Kurreck y sus colegas dicen que los resultados mostraron claramente que siCoV6 inhibe la replicación del SARS ‐ CoV ‐ 2 y protege a las células de la toxicidad celular inducida por infecciones.
Analizando más a fondo los efectos de siCoV6 en variantes
Dado que el SARS ‐ CoV ‐ 2 está en constante evolución y las mutaciones pueden resultar en la alteración de las secuencias diana de siRNA, el equipo analizó alrededor de 1500 de las secuencias más recientes de diferentes variantes del SARS ‐ CoV ‐ 2 con respecto a las mutaciones en el sitio diana de siCoV6.
Los investigadores no identificaron ninguna mutación en el sitio objetivo de la variante B.1.351 (beta) o B.1.617.2 (delta), y para las variantes alfa y P.1 (gamma), solo menos del 0,8% de las secuencias. tenía mutaciones en el sitio diana siCoV6.
Kurreck y sus colegas dicen que este trabajo ha identificado un ARNip muy potente con una amplia actividad contra varios virus del SARS-CoV que representa un candidato prometedor para el desarrollo de nuevas opciones de tratamiento de COVID-19.